5 Assoziationsmuster
Wir möchten nun zusätzlich wissen, welche Lexeme überzufällig häufig als Bestimmungsglieder in Corona-Komposita auftreten. Dafür brauchen wir zunächst ein Referenzkorpus. Hierfür habe ich eine Frequenzliste aller als Nomen getaggten Tokens aus dem DWDS-Kernkorpus des 21. Jahrhunderts erstellt (wiederum mit Dstar), die wir nun einlesen. Wiederum konvertieren wir alle Daten in Kleinschreibung und zählen die Frequenz entsprechend aus:
# Daten einlesen
nouns <- read_delim("allnouns_dwds21.txt",
delim = "\t", quote="",
col_names = c("Freq_dwds21", "word"))
# alle in Kleinschreibung
nouns$word <- tolower(nouns$word)
# neu auszählen (um groß- und kleingeschriebene Varianten zu vereinen)
nouns <- nouns %>% group_by(word) %>% summarise(
Freq_dwds21 = sum(Freq_dwds21)
)Nun verbinden wir mit Hilfe der left_join-Funktion die weiter oben erstellte corona_tbl-Tabelle mit der soeben erstellten nouns-Tabelle. Da nicht alle Lexeme, die in der Corona-Tabelle belegt sind, auch in der DWDS21-Tabelle belegt sind, gibt es einige fehlende Datenpunkte (in R heißen diese NA, für “Not Available”). Diese ersetzen wir mit Hilfe des replace_na-Befehls durch 0.
corona_tbl <- left_join(corona_tbl, nouns, by = c("head" = "word"))
corona_tbl <- replace_na(corona_tbl, list(Freq_dwds21 = 0))Mit Hilfe der Funktion collex.dist aus dem “collostructions”-Paket (Flach 2017) können wir nun eine sog. distinktive Kollexemanalyse über die Daten laufen lassen. Wenn Sie mehr über dieses Verfahren lesen möchten, können Sie z.B. das Paper von Gries & Stefanowitsch (2004) oder den Überblicksartikel zur Kollostruktionsanalyse von Stefanowitsch (2013) lesen. Empfehlenswert sind auch die Einführungsvideos von Susanne Flach (sfla.ch), wo Sie auch eine Einführung in das gleichnamige R-Paket finden. Beachten Sie aber bitte, dass ich dieses Verfahren hier überhaupt nicht im Sinne der Erfinder verwende, sondern lediglich explorativ auf Datensätze anwende. Deshalb vermeide ich im Folgenden auch Termini wie “Kollostruktionsanalyse” oder “Kollexemanalyse”, sondern spreche allgemein von “Assoziationsmustern”. Die Kollostruktionsanalyse habe ich nur erwähnt, weil das besagte Paket eine praktische Möglichkeit bietet, auf einfachste Weise Assoziationsmuster zu errechnen, und der Name des Pakets andernfalls für Uneingeweihte etwas kryptisch bleiben würde. - Wenn Sie mehr darüber erfahren möchten, welche text- und diskurslinguistischen Aufschlüsse Assoziationsmuster im Allgemeinen erlauben, empfehle ich Bubenhofer (2009) oder auch viele der Blogeinträge, die über Bubenhofers Website verfügbar sind (https://www.bubenhofer.com/).
Wir führen nun die Kollostruktionsanalyse durch und schauen uns die ersten paar Einträge an, die uns der head-Befehl zeigt:
corona_coll <- collex.dist(as.data.frame(corona_tbl))
# Ich stelle hier die Tabelle ohne die Spalte SHARED dar (die angibt, ob ein Lemma in beiden Datensätzen vorkommt), damit sie in eine Zeile passt:
select(corona_coll, -SHARED) %>% head(10)## COLLEX O.CXN1 E.CXN1 O.CXN2 E.CXN2 ASSOC COLL.STR.LOGL SIGNIF
## 1 virus 32628 6623.4 237 26241.6 Freq 112753.565 *****
## 2 krise 18019 3715.5 417 14720.5 Freq 56986.655 *****
## 3 pandemie 6188 1247.1 0 4940.9 Freq 20168.335 *****
## 4 viruspandemie 2221 447.6 0 1773.4 Freq 7158.780 *****
## 5 infektion 1362 281.5 35 1115.5 Freq 4067.988 *****
## 6 viruskrise 1076 216.9 0 859.1 Freq 3457.251 *****
## 7 virusinfektion 1044 211.4 5 837.6 Freq 3292.928 *****
## 8 patient 1027 228.7 108 906.3 Freq 2634.028 *****
## 9 epidemie 759 156.4 17 619.6 Freq 2280.648 *****
## 10 virusfall 675 136.0 0 539.0 Freq 2166.428 *****Erwartungsgemäß führen -virus und -krise die Rangliste an, gefolgt von -pandemie und patient. Hier also keine Überraschungen. Interessant kann es auch sein, sich die letzten Ränge anzuschauen:
## COLLEX O.CXN1 E.CXN1 O.CXN2 E.CXN2 ASSOC COLL.STR.LOGL
## 2754 weg 1 770.5 3822 3052.5 Freq_dwds21 1713.260
## 2755 preis 1 809.4 4015 3206.6 Freq_dwds21 1800.886
## 2756 daten 30 996.0 4912 3946.0 Freq_dwds21 1953.955
## 2757 version 1 924.0 4584 3661.0 Freq_dwds21 2059.496
## 2758 deutschland 13 986.5 4882 3908.5 Freq_dwds21 2072.096
## 2759 seite 3 954.7 4734 3782.3 Freq_dwds21 2102.843
## 2760 leben 2 964.9 4786 3823.1 Freq_dwds21 2138.409
## 2761 jahr 14 1066.5 5278 4225.5 Freq_dwds21 2241.806
## 2762 welt 11 1073.6 5316 4253.4 Freq_dwds21 2285.740
## 2763 programm 10 1259.2 6238 4988.8 Freq_dwds21 2712.821
## SIGNIF
## 2754 *****
## 2755 *****
## 2756 *****
## 2757 *****
## 2758 *****
## 2759 *****
## 2760 *****
## 2761 *****
## 2762 *****
## 2763 *****Wörter Deutschland, Seite, Leben, Welt kommen also relativ selten als Bestimmungsglied von Corona-Komposita vor.
Wir haben nun auch die Möglichkeit, diese Assoziationswerte in unser oben erstelltes MotionChart zu integrieren, um so auch auf der x-Achse einen (halbwegs) bedeutungsvollen Wert zu zeigen. Weil die Assoziationswerte ebenfalls eine sehr schiefe Verteilung aufweisen, ist es sinnvoll, sie ebenfalls zu logarithmieren - das ist aber wirklich nur in diesem speziellen Fall sinnvoll, normalerweise würde man das eher nicht machen. Auch ist es so, dass die Assoziationen in unterschiedliche Richtungen gehen: Manche haben einen hohen LogLikelihood-Wert (das ist das Assoziationsmaß, das verwendet wurde), weil sie überzufällig häufig in den Corona-Daten auftauchen, andere haben einen hohen LogLikelihood-Wert, weil sie überzufällig häufig im Referenzkorpus auftauchen. Daher füge ich noch eine zusätzliche Spalte hinzu, in der ich diejenigen Werte, bei denen die Tendenz in Richtung Referenzkorpus (und damit weg vom Corona-Korpus) geht, negativiere.
corona_coll$Log_assoc <- log(corona_coll$COLL.STR.LOGL)
corona_coll$Log_assoc2 <- ifelse(corona_coll$ASSOC=="Freq_dwds21", -corona_coll$Log_assoc, corona_coll$Log_assoc)Um die gewünschte Visualisierung erhalten zu können, müssen wir die Information zur Assoziationsstärke zur oben erstellten d2-Tabelle hinzufügen, mit der wir das MotionChart auf der vorigen Seite erstellt haben. Dafür nutzen wir wieder die left_join-Funktion. Mit dem by-Argument geben wir an, dass die Spalte mit den Daten, die in beiden Tabellen vorkommen und damit quasi als “Scharnier” für die Kombination beider Tabellen dienen, in der ersten Tabelle einen anderen Namen hat als in der zweiten; in d2 heißt sie nämlich “head” und in corona_coll “COLLEX”. Die Spaltennamen in der corona_coll-Tabelle sind alle die Default-Spaltennamen, die der Output der Funktionen im “collostructions”-Paket aufweist - nur falls Sie sich gewundert haben sollten, wo dieser Spaltenname herkommt.
Die Daten können wir nun in bewährter Manier visualisieren.
bubble2 <- gvisMotionChart(d2, idvar = "head", timevar = "date",
xvar = "Log_assoc2", yvar = "logFreq",
sizevar="logFreq")
plot(bubble2)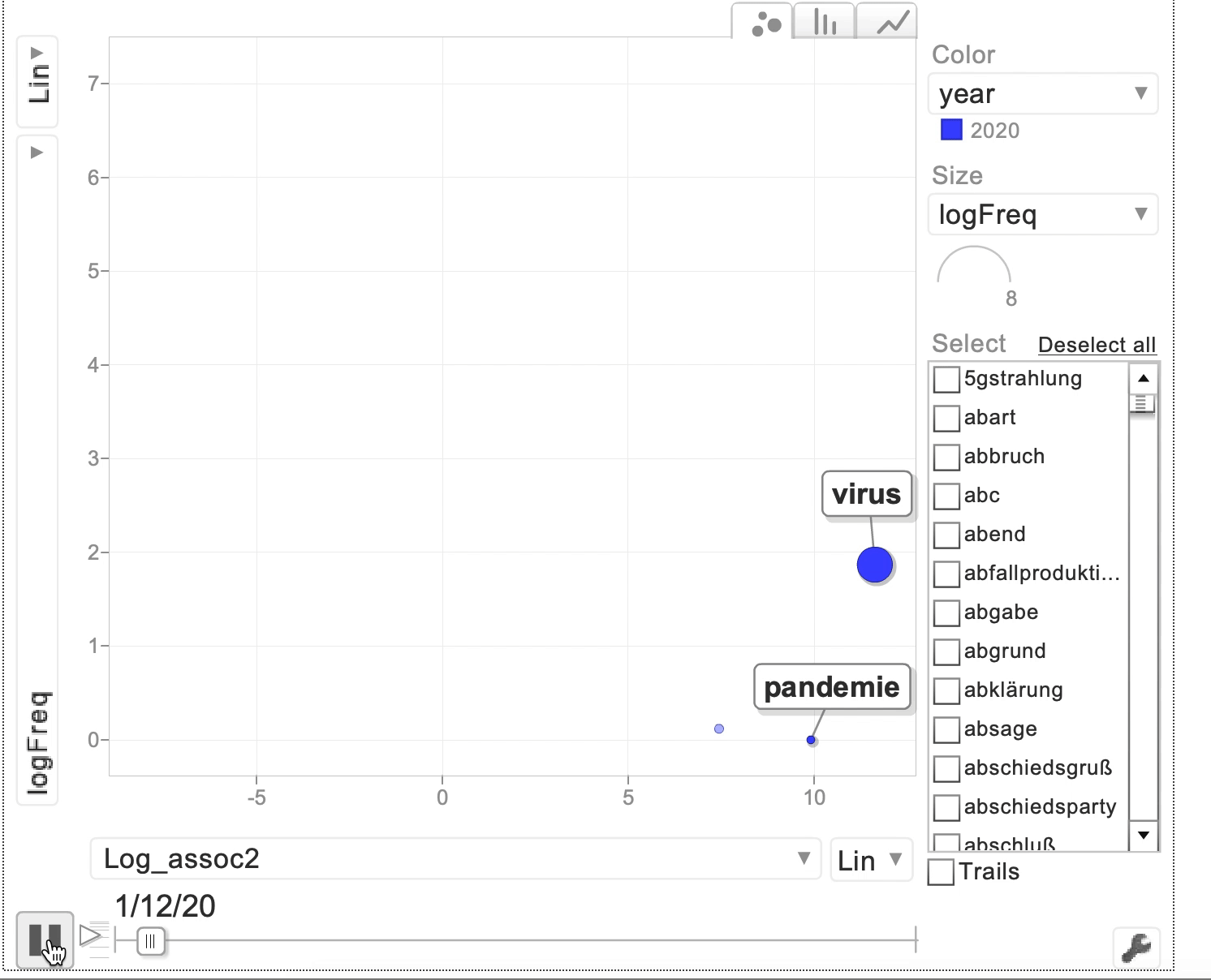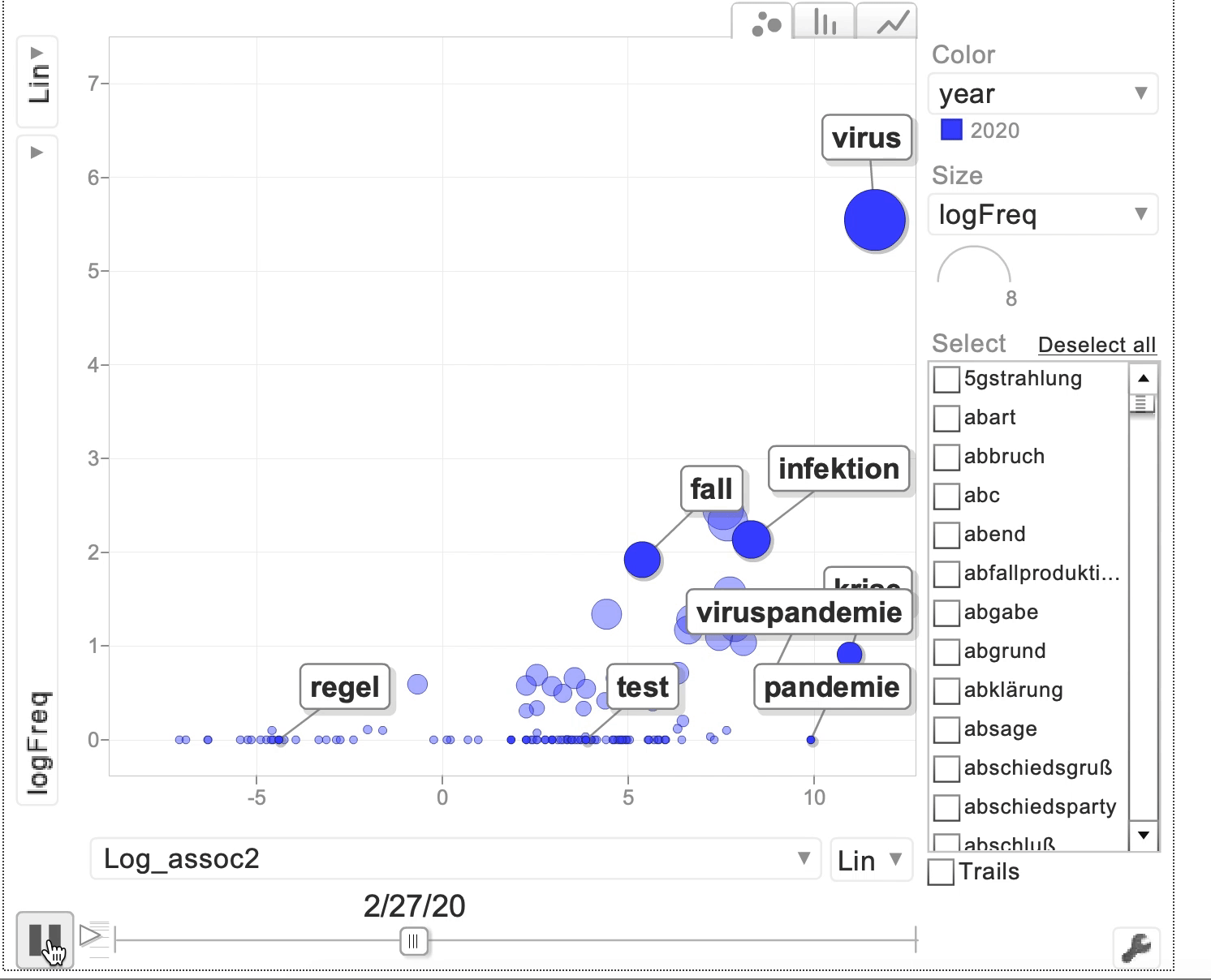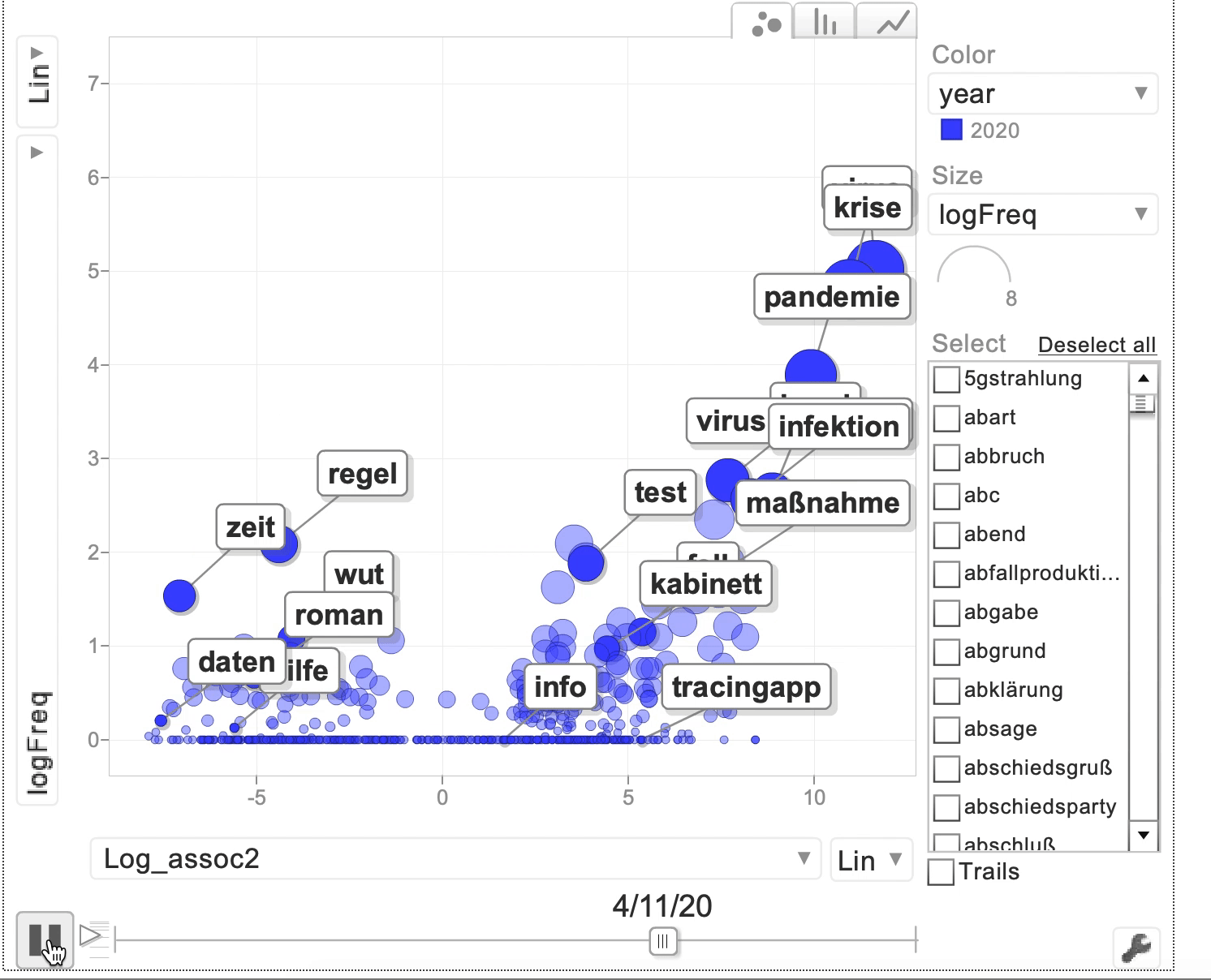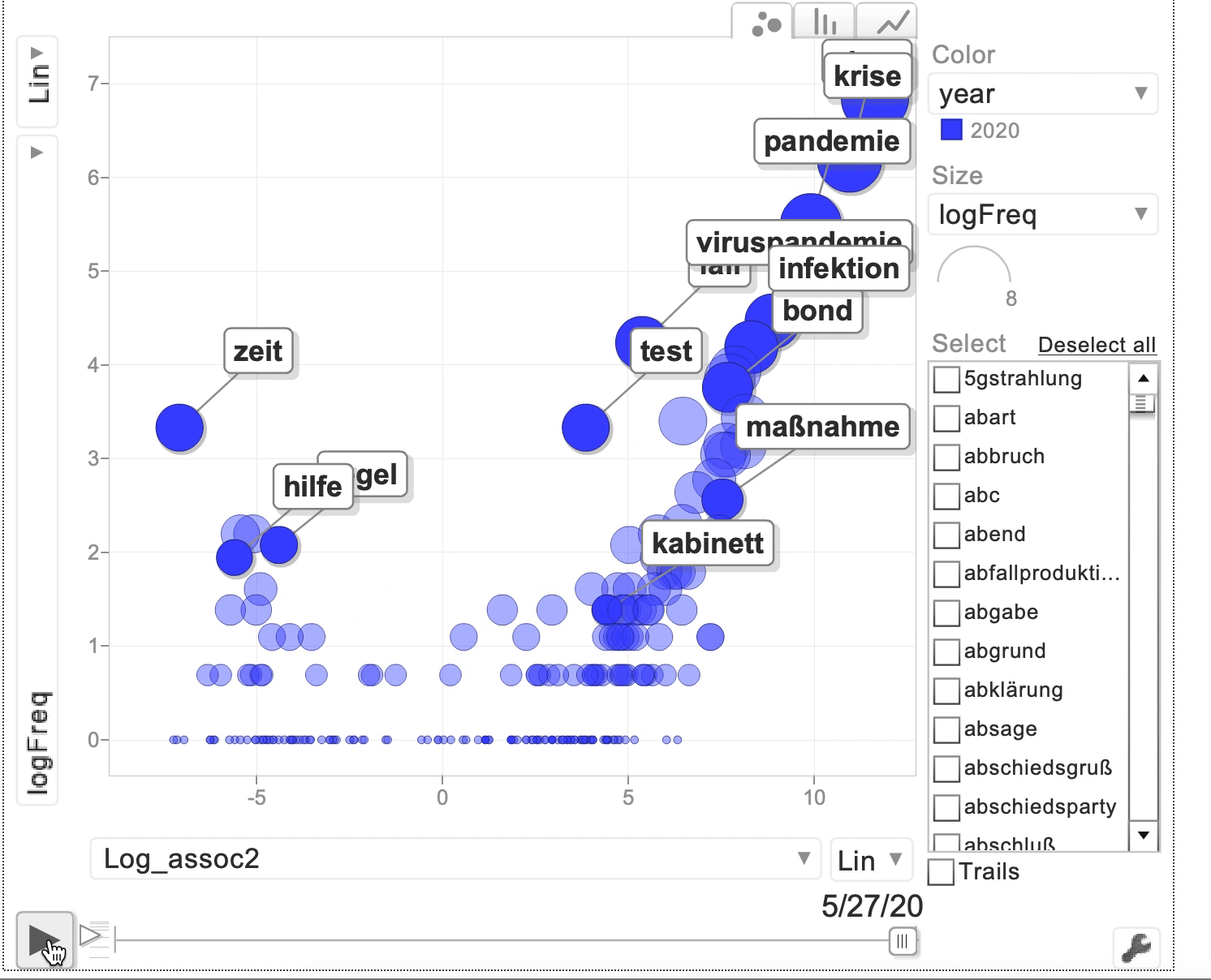
Fig. 5.1: GoogleVis Motion Chart mit Frequenzen der Bestimmungsglieder, die im Coronakorpus mit dem Erstglied Corona- auftreten.
Statt eines Schlussworts eher eine Zwischenbilanz: Ich hoffe, Sie konnten das eine oder andere aus diesem Tutorial lernen, auch wenn es noch work-in-progress ist und gerade die letzten beiden Seiten noch etwas Feinschliff bedürften, den ich hoffentlich bald ergänzen kann. Was aber deutlich geworden sein dürfte, ist, dass wir gerade aus der explorativen Auswertung der Daten interessante Trends ablesen können, auch wenn an der einen oder anderen Stelle Vorsicht geboten ist, gerade was die Genauigkeit der Daten angeht (Coronaauflagen erscheinen in den Daten zum Beispiel verdächtig früh, was darauf hindeutet, dass evtl. einige Daten nicht ganz stimmen). Anhand der Daten konnten wir uns zudem einige Grundbegriffe aus der korpusbasierten Erforschung morphologischer Phänomene erschließen, auch wenn wir mit den konkreten Kenngrößen kontextfrei nicht allzu viel anfangen könnten, sondern hierfür Vergleichswerte bräuchten, um interessante Tendenzen und Beobachtungen daraus ableiten zu können.